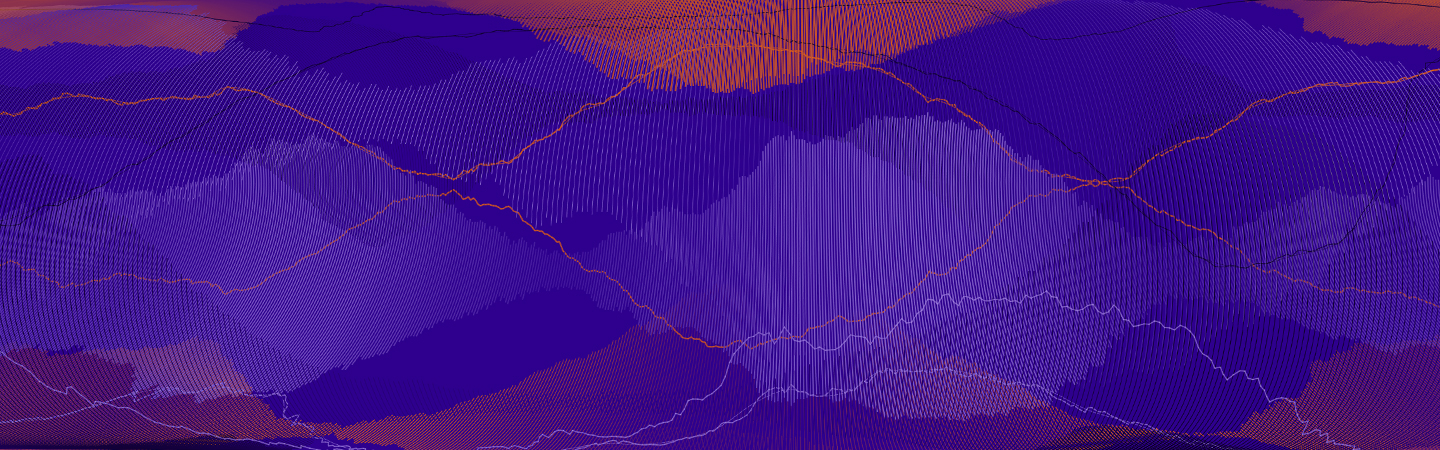
Die Datenwissenschaften sind mit ihren langen Zahlenkolonnen, Programmcodes und Formeln oft sehr abstrakt. Mit dem Projekt "Der Zauber der Daten" haben wir versucht, die Daten, mit denen unsere Promovierenden arbeiten, sinnlich und greifbar darzustellen.
Daten in neuem Gewand
Immer mehr Wissenschaftlerinnen und Wissenschaftler arbeiten heutzutage mit Datensätzen, die nicht nur äußerst umfangreich sind, sondern ihre Forschungsgegenstände auf eine sehr abstrakte Weise darstellen. Diese Zahlenreihen sind für Außenstehende schwer zu verstehen, so dass die Faszination, die von Daten ausgehen kann, oft nur denjenigen zugänglich ist, die mit ihnen forschen. HIDA hat versucht, dem komplexen Thema Data Science eine visuelle Form zu geben, um es sinnlich erlebbar zu machen. Das Ziel: Über die Bilder einen Zugang zur modernen datengetriebenen Forschung zu schaffen.

Der Zauber der Daten – HIDAs Datenanimationen für den re:publica Campus 2020 in Berlin
Die hier gezeigten Animationen und Visualisierungen sind künstlerische Arbeiten, die der Designer Michael Schmitz auf Basis von echten Datensätzen erstellt hat. Mit ihnen forschen Doktorandinnen und Doktoranden der sechs Helmholtz Data Science Schools zu Themen rund um Energie, Erde und Umwelt, Gesundheit, Schlüsseltechnologien, Struktur der Materie sowie Luftfahrt, Raumfahrt und Verkehr.
Aus Daten werden Bilder
Wie werden aus „unsichtbaren“ Zahlenwerten eindrucksvolle und unverwechselbare künstlerische Arbeiten?
Der Gestaltungsprozess beginnt mit einer vagen Idee, erzählt Michael Schmitz. Am Anfang seiner Arbeit schreibt der Designer einen Algorithmus, der wie ein Regelwerk funktioniert und die Datenpunkte, die zunächst nur als Zahlenwerte in langen Excel-Listen vorliegen, mithilfe des Computerprogramms Processing in grafische Elemente „übersetzt“ - etwa in Koordinaten, Radien, Strichstärken oder Bogenmaße. Wie das konkrete Ergebnis aussieht, sobald ein Datensatz durch den Algorithmus läuft, bleibt aber zunächst offen, sagt Schmitz.
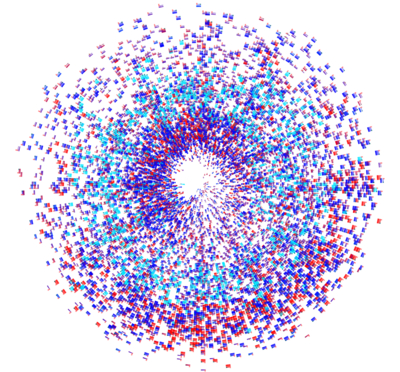
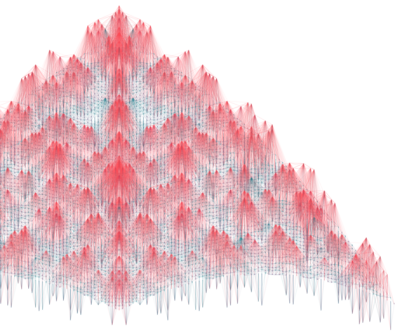
Generatives Design
Diese sogenannten generativen Grafiken sind aber noch weit mehr als die künstlerische Übersetzung von Daten, denn sie verändern auch die Arbeitsweise von Künstlern auf eine fundamentale Art und Weise:
Bisher galt, dass der Künstler als Genie sein Bild oder seine Skulptur kontrolliert formt und die Details nach seiner Vorstellung gestaltet. Mit den generativen Grafiken wird der Künstler zum Programmierer eines Regelwerkes. Er bestimmt nur die Parameter für die Erstellung seines Objekts. Nach diesen festgelegten Definitionen generiert die Software ein nicht vorhersehbares Bild. Am Ende wählt der Künstler zwar die gelungenen Ergebnisse als kreativen Akt aus, aber das Kunstwerk selbst ist das Resultat eines autonomen Prozesses. Der Künstler wird so zum Schöpfer, der den Rahmen und die Normen setzt, innerhalb derer unvorhersehbare Kunstwerke errechnet werden.
Die Forschung hinter den Datenbildern

Arktischer Permafrost
Auftau-Dynamik des arktischen Permafrosts
Tabea Rettelbach von der HEIBRiDS nutzt in ihrem Forschungsprojekt Methoden des maschinellen Lernens, um anhand von hochaufgelösten Erdbeobachtungsdaten die Auftau-Dynamik des arktischen Permafrosts zu erkennen.
Der für diese Darstellung verwendete Datensatz beschreibt die Länge und die Lage von ca. 3 000 Eiskeil-Spalten in der Arktis Alaskas im Juli 2019.
Eiskeile sind ein weit verbreitetes Phänomen arktischer Permafrost-Landschaften. Sie entstehen durch das Gefrieren und Aufreißen der Böden im Winter. Im Frühjahr dringt Schmelzwasser in diese tiefen Risse und gefriert wieder im Dauerfrost des Bodens. Über Jahrtausende bilden sich auf diese Weise keilförmige, massive Eiskörper, die weite, regelmäßige Strukturen in der Landschaft bilden und von oben betrachtet wie Trockenrisse aussehen.
Diese eisgefüllten Bodenspalten werden sehr genau beobachtet (u. a. durch Luft- und Satellitenbilder), um Aussagen über die klimatischen Veränderungen, deren Folgen für die arktische Permafrost-Landschaft und die dadurch beschleunigte Freisetzung von Kohlendioxid aus den aufgetauten Böden machen zu können.


Innere Körper-Uhr
Kontrollmechanismen unserer Körper-Uhr
Vojtech Kumpost von der HIDSS4Health versucht mit mathematischen Modellierungen zu verstehen, wie biologische Kontrollmechanismen funktionieren, die unsere innere Körper-Uhr steuern.
Die Darstellung beruht auf den Daten eines simulierten zyklischen Schwingungsverhaltens von drei unterschiedlichen Proteinen. Die Zeit ist in Stunden, die Konzentration in willkürlichen Einheiten angegeben.
Unser Körper hat eine innere Uhr, damit er weiß, wann es Zeit ist zu schlafen, zu essen oder aktiv zu sein. Diese innere Uhr kann auch auf der Ebene einzelner Zellen beobachtet werden, wo die Proteinkonzentrationen synchron mit dem 24-Stunden-Tag-Nacht-Zyklus schwanken.
Die hier abgebildeten Daten wurden durch ein mathematisches Modell erzeugt, das dieses Schwingungsverhalten („zyklische Oszillation“) beschreibt. Diese Daten werden verwendet, um die Kontrollmechanismen besser zu verstehen, die unsere innere Uhr im richtigen Tempo ticken lassen. Die Untersuchung dieser Mechanismen ist wichtig, weil jede Störung der inneren Uhr (z.B. verursacht durch Jetlag, Schichtarbeit oder Genmutationen) zu einer geringeren Lebensqualität und einer Verschlechterung des Gesundheitszustands führt (z.B. schlechte Schlafqualität, Depression und Diabetes).


Gehirn-Aktivität 1
Synchrone Gehirn-Aktivität
Christian Gerloff von der HDS-LEE überträgt Methoden der künstlichen Intelligenz und des Maschinenlernens auf die Neurowissenschaften, um zu einem besseren Verständnis des menschlichen Gehirns zu gelangen.
Der Datensatz, auf dem diese Darstellung beruht, enthält Signale aus 22 verschiedenen Regionen des menschlichen Gehirns. Sie stammen jeweils von zwei interagierenden Versuchs-Teilnehmern. Festgehalten sind die Hirnregion, die Quelle, die Messpunkt-Koordinaten sowie die Änderungsrate der Oxyhämoglobin-Konzentration.
Die Daten wurden gleichzeitig erhoben und geben Auskunft über die Oxyhämoglobin-Konzentration in den einzelnen Hirnarealen der Probanden. Sie wird mittels funktioneller Nahinfrarotspektroskopie (fNIRS) erhoben: Dabei wird durch die Kopfhaut der Probanden Infrarotlicht gestrahlt, dessen Reflektion wiederum gemessen wird. Damit lässt sich erkennen, wohin das Blut im Gehirn geschickt wird – vermutlich, weil die Neuronen in diesem Bereich während einer geistigen Aufgabe aktiver sind. Anhand solcher Versuche erhofft man sich Erkenntnisse darüber, wie zwei Gehirne, die eine Aufgabe gemeinsam lösen, kooperieren.
Die hier gezeigte Darstellung und die nachfolgende Abbildung zeigen, wie unterschiedlich die Visualisierungen ausfallen können, je nachdem, wie der ihnen zugrunde liegende Algorithmus die zur Verfügung stehenden Formelemente auswählt: Obwohl die eingespeisten Daten identisch sind, werden sie durch den Algorithmus in verschiedene grafische Elemente „übersetzt“.


Gehirn-Aktivität 2
Synchrone Gehirn-Aktivität
CHRISTIAN GERLOFF von der HDS-LEE überträgt Methoden der künstlichen Intelligenz und des Maschinenlernens auf die Neurowissenschaften, um zu einem besseren Verständnis des menschlichen Gehirns zu gelangen.
Der Datensatz, auf dem diese Darstellung beruht, enthält Signale aus 22 verschiedenen Regionen des menschlichen Gehirns. Sie stammen jeweils von zwei interagierenden Versuchs-Teilnehmern. Festgehalten sind die Hirnregion, die Quelle, die Messpunkt-Koordinaten sowie die Änderungsrate der Oxyhämoglobin-Konzentration.
Die Daten wurden gleichzeitig erhoben und geben Auskunft über die Oxyhämoglobin-Konzentration in den einzelnen Hirnarealen der Probanden. Sie wird mittels funktioneller Nahinfrarotspektroskopie (fNIRS) erhoben: Dabei wird durch die Kopfhaut der Probanden Infrarotlicht gestrahlt, dessen Reflektion wiederum gemessen wird. Damit lässt sich erkennen, wohin das Blut im Gehirn geschickt wird – vermutlich, weil die Neuronen in diesem Bereich während einer geistigen Aufgabe aktiver sind. Anhand solcher Versuche erhofft man sich Erkenntnisse darüber, wie zwei Gehirne, die eine Aufgabe gemeinsam lösen, kooperieren.
Die hier gezeigte Darstellung und die vorangehende Abbildung zeigen, wie unterschiedlich die Visualisierungen ausfallen können, je nachdem, wie der ihnen zugrunde liegende Algorithmus die zur Verfügung stehenden Formelemente auswählt: Obwohl die eingespeisten Daten identisch sind, werden sie durch den Algorithmus in verschiedene grafische Elemente „übersetzt“.

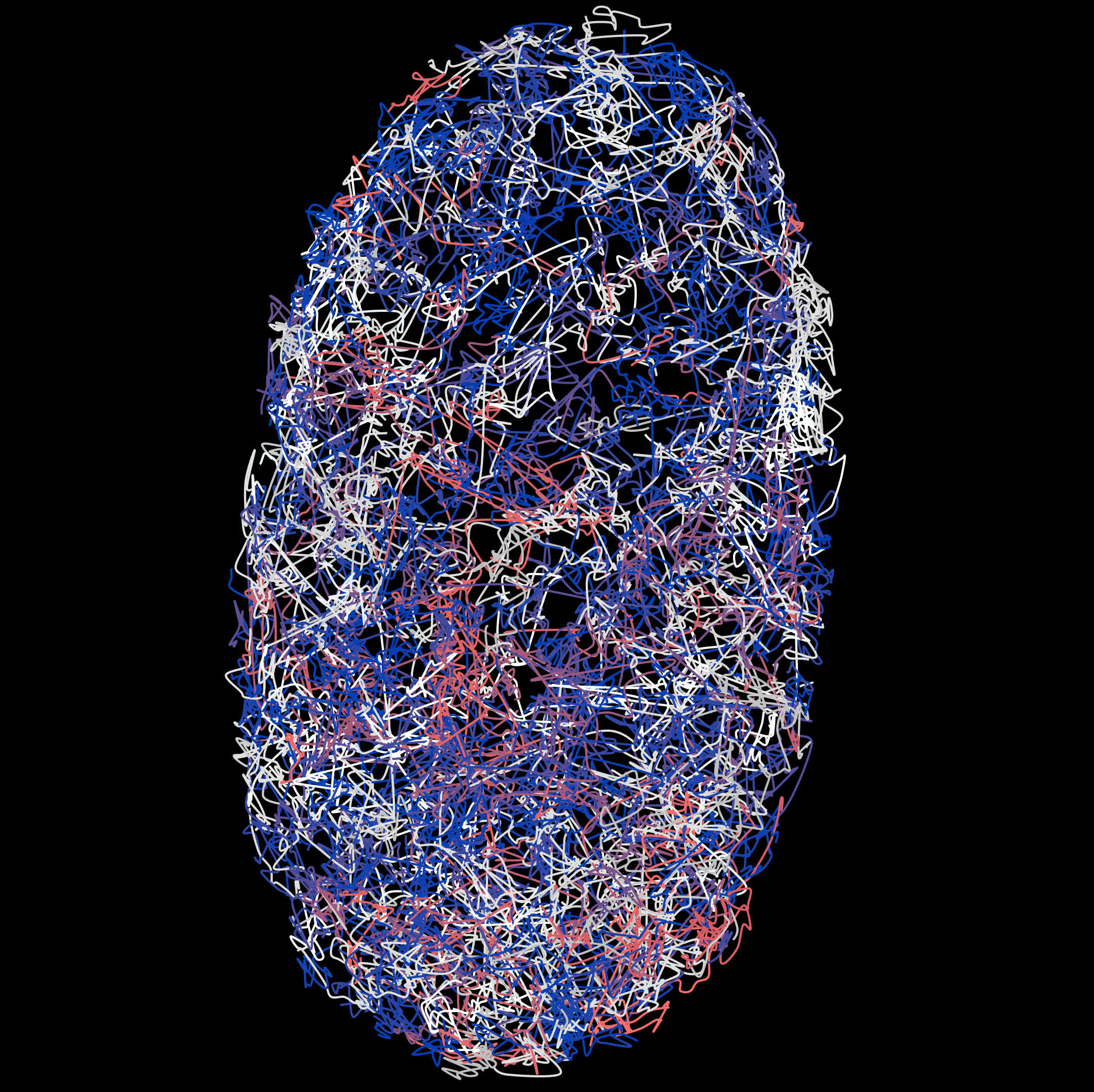
Zellkern-Bewegung
Zellkern-Bewegung
Katharina Löffler von der HIDSS4Health möchte mit Methoden des maschinellen Lernens berechnen, wie sich Zellkerne bewegen, um genauere Kenntnisse über die Entwicklung von Organen oder die Entstehung von Krankheiten zu ermöglichen.
Die hier verwendeten Daten umfassen die Positionen der Zellzentren (z, y, x) der verfolgten Zellen über die Zeit. Betrachtet wurden Zellen des Fadenwurms C. elegans.
Viele biomedizinische Bildgebungsgeräte sind in der Lage, hochauflösende 3D-Daten im Zeitverlauf zu erfassen (3D+t, t = time) – mit ihnen lassen sich z.B. Zellen über die Zeit verfolgen. Hierfür werden diese mit fluoreszierenden Stoffen markiert, unter dem Lichtscheibenmikroskop aufgenommen und die Bewegungsdaten dieser Aufnahmen anschließend mithilfe von Algorithmen analysiert.
Übliche Methoden der Bildgebung, die etwa für die Identifizierung von Zellkernen verwendet werden, haben Genauigkeiten um 95 %. Das klingt zunächst gut. Dennoch ist dies immer noch nicht ausreichend, um eine gesamte zelluläre Abstammungslinie (bestehend aus allen Zellkernbewegungen und Zellteilungen, die eine Zelle im Laufe ihrer Entwicklung durchmacht) fehlerfrei zu rekonstruieren. Um besser zu verstehen, wie sich Zellen differenzieren und die verschiedenen Organe in einem Organismus anlegen, ist eine genauere Kenntnis dieser Bewegungen hilfreich. Ziel des Projekts ist daher, 3D+t-Verfolgungsalgorithmen zu verbessern.
Die hier gesammelten Daten zielen auf die quantitative Beschreibung von Zelltod, Zellteilung, Bewegung und Interaktion ab – betrachtet am Beispiel der Entwicklung des Fadenwurms C. elegans, eines beliebten Modellorganismus’.
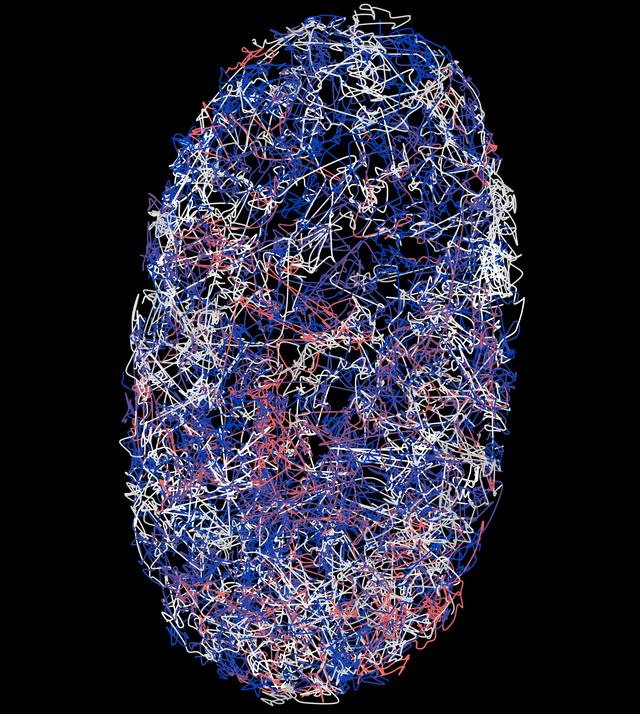

Röntgendaten-Analyse
Röntgendaten-Analyse
Stasis Chuchurka von der DASHH forscht mit datenwissenschaftlichen Methoden daran, ein einheitliches Modell für die Analyse von Röntgendaten zu entwickeln, um die elektronischen Strukturen von Stoffen und folglich chemische Prozesse besser zu verstehen.
Die für diese Darstellung verwendeten Daten beziehen sich auf die elektronische Ladungsdichte von Harnstoff (Urea). Sie umfassen die Dichtematrixelemente und je zwei dazu korrespondierende Atomorbitale.
Die Eigenschaften von chemischen Stoffen werden stark durch ihre elektronische Struktur bestimmt. Subtile Veränderungen in der elektronischen Struktur sind von großer Bedeutung für das Verständnis chemischer Prozesse, etwa der Photosynthese oder der Katalyse. Aktuelle Simulationen dieser Struktur-Veränderungen liefern aber keine akzeptable Genauigkeit und erfordern viel Rechenzeit. Andererseits geben experimentelle Daten allein auch keine vollständige Information über die Elektronenstruktur.
Um dieses Problem zu lösen, müssen experimentelle und theoretische Ansätze auf systematische Weise kombiniert werden. Das Forschungsprojekt ist daher interdisziplinär zwischen den Naturwissenschaften und der Mathematik angesiedelt, um mit datenwissenschaftlichen Rechenmethoden ein neuartiges und einheitliches Modell der Röntgendatenanalyse zu entwickeln. Dies soll Daten aus unterschiedlichen experimentellen Ansätzen kombinieren, die sonst nur getrennt analysiert werden können, z.B. Daten aus Röntgenbeugungs- und Röntgenemissionsspektroskopie-Messungen.
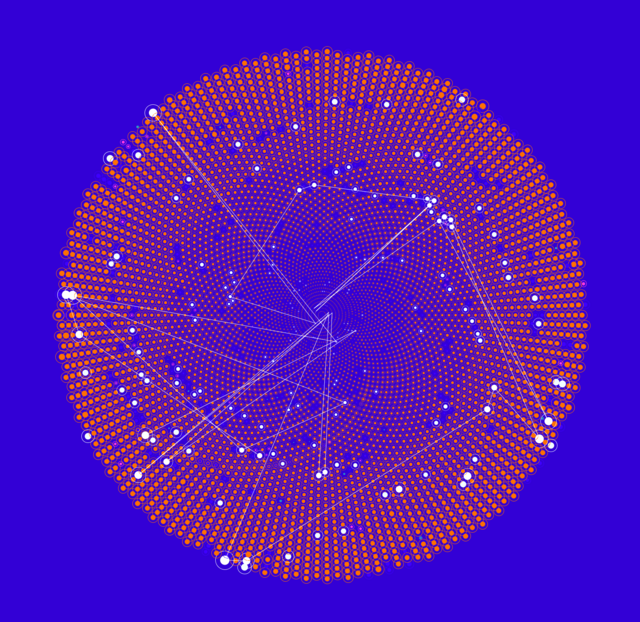

Stoffwechsel-Prozesse
Stoffwechsel-Prozesse
Johann Fredrik Jadebeck von der HDS-LEE arbeitet an computer-basierten Methoden, um besser zu verstehen, mit welcher Geschwindigkeit Stoffwechselvorgänge in Mikroorganismen ablaufen.
Die Daten stellen die Häufigkeits-Verteilungen (basierend auf einem repräsentativen Stichproben-Verfahren) einzelner Stoffwechsel-Reaktionsraten (Flüsse) im Bakterium Escherichia colidar.
Der Stoffwechsel (Metabolismus) beschreibt die biochemischen Vorgänge, die im Inneren von Zellen vor sich gehen: Wie gewinnen Zellen aus Nährstoffen Energie, um zu wachsen? Wie leiten sie Stoffströme, um bestimmte Substanzen herzustellen? Ungewiss ist, mit welcher Geschwindigkeit (kurz „Fluss“ genannt) diese Reaktionen ablaufen. Biotechnologen sind an Flüssen in lebenden Zellen interessiert, da sie Rückschlüsse darauf zulassen, wie sich z. B. eine bessere Ausbeute eines Substrats erzielen lässt. Auch für die Medizin sind Reaktionsstärken interessant, etwa in der Wirkstoffforschung: Wie unterscheiden sich die Flüsse gesunder und kranker Zellen? Und wie wirkt sich ein Medikament auf sie aus?
Die metabolischen Reaktionsraten im Zellinneren von Mikroorganismen kann man jedoch nicht direkt messen. Hier helfen indirekte Informationen aus Kohlenstoff-Markierungsexperimenten weiter: Hierzu werden Moleküle, die die Organismen aufnehmen, vorher „markiert“, so dass sich anhand der Konzentration der spezifischen Markierungsmuster dieser Moleküle in den Zellen Rückschlüsse über den erfolgten Stoffwechselvorgang treffen lassen. Mit Computermodellen lassen sich aus diesen Messergebnissen die Flüsse ermitteln. Datenwissenschaftliche Rechenmethoden helfen dabei, diese noch genauer zu bestimmen.


Selbst-Navigation
Selbst-Navigation
Felix Fiedler von der HEIBRiDS will mit Methoden der künstlichen Intelligenz die komplexe Datenanalyse für Selbstlokalisierungssysteme energiearm und leistungsstark gestalten.
Die für diese grafische Umsetzung verwendeten Daten umfassen Längen- und Breitengrade, Zeitangaben und Geschwindigkeit.
Die Fähigkeit zur Selbstlokalisierung ist für alle Aufgaben entscheidend, bei denen autonome Entscheidungen getroffen werden müssen, z. B. von Roboterfahrzeugen auf planetarischer Erkundungsmission. Ein Navigationssystem, das dies leisten kann, benötigt keine externe Referenz wie Satellitendaten, um einen Punkt im Raum zu bestimmen.
Diese Darstellung beruht auf den Daten, die ein solches Navigieren möglich machen sollen: Eine Person, ein Fahrzeug oder ähnliches ist mit einem Gerät ausgestattet, das versucht, auf die relative Position (bezüglich des Startpunkts) und auf die Geschwindigkeit zu schließen. Daten von verschiedenen Sensoren (Kamera und Beschleunigungsmesser) werden zusammengeführt und berechnet, um die plausibelste Vorhersage zu treffen. Ziel ist es, dass künftige Navigationsgeräte diese rechnerische „Fusion“ von Sensorinformationen mit höchster Genauigkeit und energiearm durchführen. Hierfür werden neue Algorithmen und Techniken des maschinellen Lernens entwickelt.

Ozon-Konzentration
Ozon-Konzentration
Helge Mohn von der MarDATA will mithilfe von neuronalen Netzen Veränderungen der Ozonkonzentration in der Atmosphäre mit geringem Rechenaufwand und hoher Genauigkeit bestimmen, damit Klimamodelle bessere Voraussagen treffen können.
Die Darstellungen beruhen auf Daten, mit denen die Ozon-Änderungsraten berechnet werden. Der zeitliche Input ist der Tag des Jahres, die räumliche Information ist durch den Breitengrad und die Höhe gegeben. Hinzu kommen verschiedene andere Parameter (Sonnenenergie, Temperatur und die chemische Konzentration verschiedener Stoffverbindungen von Chlor, Brom, Stickstoffoxid und Wasserstoff).
Aktuelle chemische Modelle in der Erdsystem-Modellierung können physikalische und chemische Prozesse detailliert darstellen. Sie enthalten Hunderte von chemischen Stoffen und Reaktionen, die in komplexe Gleichungen integriert sind. Für deren Lösung sind oft große Rechencluster erforderlich. Will man solche chemischen Modelle nun mit Klimamodellen koppeln – etwa um die Effekte von Änderungen in der chemischen Zusammensetzung der Atmosphäre auf das Klima vorherzusagen –, ist dies nur möglich, wenn die Rechenzeit erheblich erhöht wird. Die Einbeziehung detaillierter Chemie in Klimamodelle ist jedoch wünschenswert, um die verschiedenen Rückkopplungen zwischen chemischen Prozessen, der Atmosphäre und insbesondere dem Ozean zu berücksichtigen. Die Konzentration von Ozon als ein besonders klimarelevantes Atmosphärengas in der Stratosphäre ist dabei ein wichtiger Parameter.
SWIFT, ein bereits bestehendes Ozonchemie-Berechnungsschema, befasst sich mit dieser Frage. Es erlaubt bereits, eine interaktive Ozonschicht für globale Klimamodelle ohne lange Rechenzeit zu erstellen. Neuronale Netze sollen nun die bestehende Methodik bei SWIFT weiter verbessern. Sie sind besser in der Lage, nichtlinear ablaufende Änderungen in chemischen Verbindungen zu erfassen, wodurch Klimavorhersagen noch genauer werden können.

Erdbeben-Vorhersage
Erdbeben-Vorhersage
Jannes Münchmeyer von der HEIBRiDS will mit Methoden der künstlichen Intelligenz Erdbeben und ihre voraussichtliche Stärke schneller bewerten als bisher.
Die für diese grafische Umsetzung verwendeten Daten beziehen sich auf Erdbeben in Nord-Chile im Jahr 2014 und umfassen die Erdbebenstärke, Zeiteinheiten, Längen- und Breitengerade, die Tiefe in Kilometern und die Messungenauigkeit der Erdbebenstärke.
Erdbeben senden zwei grundlegende Arten von Wellen aus: schnell wandernde, aber weniger energiereiche Druckwellen und sogenannte Scherwellen, die zwar langsamer voranschreiten, dafür aber sehr energiereich sind und daher Schäden verursachen.
Die Frühwarnung funktioniert über die Einschätzung des Bebens aus den zuerst eintreffenden Druckwellen, so dass man vor den gefährlichen Scherwellen mit einem Vorlauf von bis zu 20 Sekunden warnen kann. Bisherige Algorithmen, die auf relativ wenigen Wellenmerkmalen beruhen, sind recht gut geeignet, um Erdbeben bis zu einer Momentenmagnitude von 6,5 gut abzuschätzen. Bei größeren Erdbeben ist aber die Gesamtbruchzeit des Bebens meist länger als die typischen Warnzeiten. Das bedeutet, dass die ersten schädlichen Wellen eintreffen, während das Erdbeben selbst noch fortschreitet und stärker werden kann.
Das Ziel ist, neue Algorithmen zu entwickeln, die die endgültige Bruchdauer eines Erdbebens auf der Grundlage der ersten paar Sekunden der Druckwellen und unter Berücksichtigung zusätzlicher Daten berechnen können.


Enzymstrukturen
Vorhersage von Enzymstrukturen
Karel van der Weg von der HDS-LEE nutzt neuronale Netzwerke, um Enzymstrukturen vorherzusagen.
Die hier verwendeten fünf Datensätze zeigen räumliche Strukturen ähnlicher Enzyme, die dieselbe Funktion auf verschiedene, in ihm enthaltene Aminosäuren ausüben. Die verwendeten Werte sind die Positionskoordinaten x, y, z der Aminosäuren.
Die Strukturen sind alle gleich ausgerichtet, mit nur minimaler Abweichung vom Mittelwert im Vergleich zur Referenzstruktur. Diese Unterschiede sind nur darin begründet, dass diese Enzymstrukturen unterschiedliche Metalle (Liganden) an je spezifischen Positionen (Bindungsstellen) binden. Die konkrete Struktur eines Proteins hängt von den jeweiligen chemischen Bindungen der Atome in den einzelnen Aminosäuren ab. Wenn eine chemische Veränderung zu erwarten ist (da verschiedene Atome auch unterschiedliche Bindungspartner haben), folgt auch eine Abweichung in der Struktur. Entsprechend der Enzymfunktion, bleiben bei einer Reaktion Teile der Struktur erhalten, während an anderen Stellen Abweichungen zu erwarten sind. Die Struktur lässt daher Aussagen über die Funktion der Enzyme zu.
Ziel des Forschungsprojekts ist, mithilfe von neuronalen Netzwerken die Funktion eines Enzyms aus seiner Struktur vorherzusagen. Grundlage hierfür ist ein Datensatz von ca. 15 000 generierten Enzymen, von denen das chemische Wissen über die Bindungsstellen extrahiert wird.


Strahlentherapie
Strahlentherapie
Pia Stammer von der HIDSS4Health möchte mit mathematischen Methoden die Auswirkungen von Unsicherheiten bei der Bestrahlung genauer vorhersagen, um Patienten eine Behandlung mit geringeren Nebenwirkungen zu ermöglichen.
In der vorliegenden Darstellung wurden folgende Daten verarbeitet: Die erwartete Dosisverteilung, die Varianz der Dosis in Bezug auf die möglichen Unsicherheitsquellen, die Raumkoordinaten x, y, z und die Gewebedichte an diesen Koordinaten (in Hounsfield-Einheiten).
Die Strahlentherapie ist einer der Eckpfeiler der modernen Krebsbehandlung, die bei 50% aller Patienten angewendet wird. Es handelt sich um eine hoch personalisierte Behandlung, die auf einem computergestützten Patientenmodell (basierend auf CT-Bildern) und auf einer Simulation der in den Körper des Patienten eingebrachten Strahlendosis beruht. Dies ermöglicht eine Optimierung der Behandlung vor Therapiebeginn. In der klinischen Praxis kann aber immer nur ein einziges, auf den wahrscheinlichsten Annahmen beruhendes Behandlungsszenario durchgespielt werden. Eine solche Simulation schließt jedoch nicht alle Unsicherheitsquellen ein: Neben Messfehlern bei der CT-Erfassung können etwa eine mögliche Fehlausrichtung bei der Immobilisierung des Patienten, Veränderungen in dessen Anatomie während der Behandlungssitzung (z.B. durch Atmung) oder auch zwischen den einzelnen Sitzungen (z.B. durch Gewichtsverlust) zu Ungenauigkeiten führen.
Mit modernen Methoden der mathematischen Unsicherheitsbestimmung sollen diese Abweichungen berechnet werden, um die Bestrahlung noch besser anzupassen. Auf diese Weise lassen sich negative Auswirkungen durch eine zu hohe oder zu geringe Strahlendosis minimieren.

HIDA bietet Ihnen die Datenvisualisierungen zur Nutzung an
Sie möchten HIDAs generative Datenvisualisierungen gerne verwenden? Unter diesem Link stellt HIDA Ihnen eine große Auswahl der hier präsentierten Grafiken für die private oder publizistische Nutzung als Download zur Verfügung. Hier der Überblick über die Motive. Wir bitten Sie allerdings bei der Verwendung Folgendes zu beachten:
Die Grafiken stehen unter der Creative Commons Lizenz CC BY-SA 4.0. Dies bedeutet: Die Bilder dürfen in jegliches Format kopiert und weiterverwendet werden, auch Bearbeitungen sind erlaubt. Einzige Voraussetzungen dafür sind: Die Bilder müssen mit dem Credit „Michael Schmitz für Helmholtz/HIDA“ und einem Link auf die HIDA-Website versehen werden: www.helmholtz-hida.de. Zudem müssen Bearbeitungen angegeben werden.
Kontakt

Xenia von Polier
Managerin Kommunikation & Marketing
Kontakt


Laila Oudray
Managerin Kommunikation & Marketing
Kontakt